Ciencias
Agrícolas y Veterinarias

 10.57201/IEUNA2313373
10.57201/IEUNA2313373
Sección: Artículo Original
*Autores correspondientes:
aya@biosyntech.com.py, carolina@biosyntech.com.py
Editor de área:
Andrea A.
Arrúa Alvarenga , , Universidad
Nacional de Asunción. Centro Multidisciplinario de Investigaciones Científicas
y Tecnológicas (CEMIT). San Lorenzo, Paraguay
, , Universidad
Nacional de Asunción. Centro Multidisciplinario de Investigaciones Científicas
y Tecnológicas (CEMIT). San Lorenzo, Paraguay
Editor invitado:
Guillermo
Enciso , Centro de
Desarrollo e Innovación Tecnológica (CEDIT). Hohenau, Paraguay
, Centro de
Desarrollo e Innovación Tecnológica (CEDIT). Hohenau, Paraguay
Recibido:
28
de abril de 2023
Aceptado:
19
de junio de 2023
Recibido en versión modificada:
19
de junio de 2023
Corregido:
28
de diciembre de 2023
Este
es un artículo publicado en acceso abierto bajo una Licencia Creative Commons “CC BY 4.0”. 
Declaración de conflicto: Los
autores declaran no tener conflicto de interes.
e-ISSN
2709 -0817
Como citar: Zaracho Paniagua, N. D., Maluff Ladan,
Y., Talavera Stefani, L. N., Sapper Lacy, Y. D., Prendeski Storaluk, C. E. y
Nishii Encina, E. A. (2023). Glaesserella parasuis: serotipos y
virulencia de cepas aisladas de cerdos en Itapúa-Paraguay entre febrero del
2022 a marzo del 2023. .Revista investigaciones y estudios – UNA,
14
(1), 84-89.
Glaesserella parasuis: serotipos y
virulencia de cepas aisladas de cerdos en Itapúa-Paraguay entre febrero del
2022 a marzo del 2023
Glaesserella parasuis: serotypes
and virulence of strains isolated from pigs in Itapúa-Paraguay between February
2022 and March 2023
Nadia Denisse Zaracho Paniagua1* , Yasmine
Maluff Ladan1
, Yasmine
Maluff Ladan1 , Liliana
Noelia Talavera Stefani1,2
, Liliana
Noelia Talavera Stefani1,2 , Yanina Dionisia
Sapper Lacy1
, Yanina Dionisia
Sapper Lacy1 , Carolina
Elizabeth Prendeski Storaluk1
, Carolina
Elizabeth Prendeski Storaluk1 , Eliane
Aya Nishii Encina1*
, Eliane
Aya Nishii Encina1*
1Biosyntech S. A. Área
de Investigación y desarrollo e Innovación. Fram-Paraguay.
2Universidad Nacional de
Itapúa. Facultad de Ciencias y Tecnología. Itapúa-Paraguay.
Resumen. Glaesserella
parasuis es habitualmente encontrada en el tracto respiratorio de cerdos,
aunque existen cepas asociadas a enfermedades sistémicas severas, causantes de
pérdidas económicas a nivel mundial. Existen 15 serotipos caracterizados
asociados a diferentes grados de virulencia. En Paraguay, se desconocen los
serotipos circulantes. El objetivo de este trabajo fue identificar serotipos de
G. parasuis presentes en el Departamento de Itapúa. Se incluyeron 62
cerdos con síntomas respiratorios provenientes de cinco granjas productoras de
porcinos de Itapúa durante el mes de febrero del 2022 a marzo del 2023. Los
aislados se realizaron a partir de muestras de pulmón y de cornetes colectadas
en necropsias e hisopados nasales en animales vivos. Las muestras se cultivaron
en Agar Chocolate + NAD, tras 48 hs de incubación a 37°C se realizaron pruebas
bioquímicas a las colonias sospechosas, posteriormente se realizó la extracción
de ADN a las confirmadas, seguido de PCR multiplex utilizando cebadores para
cada uno de los serotipos y un par específico de la especie. En todas las
granjas se identificó la presencia de G. parasuis, un total de 12 cepas,
entre las que se encontraban el Serotipo 2 (1), Serotipo 3 (3), Serotipo 7
(1), Serotipo 8 (1) y otras Serotipo no determinado (6), identificándose la
presencia del gen de virulencia VtaA en todas las cepas aisladas, excepto la
correspondiente al Ser8. De esta manera, este trabajo se convierte en el primer
reporte de serotipos de G. parasuis en Itapúa-Paraguay.
Palabras
clave: enfermedad de Glässer, PCR multiplex, serotipado
molecular.
Abstract.
Glaesserella parasuis
is usually found in the respiratory tract of pigs, although there are strains
associated with severe systemic diseases, causing economic losses worldwide.
There are 15 characterized serotypes associated with different degrees of
virulence. In Paraguay, the circulating serotypes are unknown. The objective of
this work was to identify G. parasuis serotypes present in the
Department of Itapúa. A total of 62 pigs with respiratory symptoms from five
pig production farms in Itapúa were included during the month of February 2022
to March 2023. The isolates were made from lung and turbinate samples collected
at necropsies and nasal swabs from live animals. The samples were cultured in
Chocolate Agar + NAD, after 48 hours of incubation at 37°C, biochemical tests were
performed on the suspected colonies, then DNA extraction was performed on the
confirmed ones, followed by multiplex PCR using specific pair of primers for
each serotype and species. In all farms, the presence of G. parasuis was
identified, on a total of 12 strains, including Serotype 2 (1), Serotype 3 (3),
Serotype 7 (1), Serotype 8 (1) and others Non-typeable (6), identifying the
presence of the VtaA virulence gene in all the isolated strains, except the one
corresponding to SV8. Thus, this work becomes the first report of G.
parasuis serotypes in Itapúa-Paraguay.
Keywords: Glässer's
disease, multiplex PCR, molecular serotyping.
Introducción
Glaesserella
parasuis puede encontrarse como comensal
colonizante del tracto respiratorio superior de los cerdos, aunque también es
un patógeno bacteriano de importancia en el sector porcino. Es agente
etiológico de la enfermedad de Glässer, y patógeno secundario del Complejo
Respiratorio Porcino (Costa-Hurtado et al., 2020; Schuwerk et al., 2020). Las
infecciones descontroladas producidas por G. parasuis resultan en
pérdidas económicas significativas en el sector porcino, causadas por
diferentes variables como atraso en el crecimiento, aumento en el índice de
conversión alimentaria, disminución en la ganancia de peso diario, costos
derivados del uso de antimicrobianos, depreciación de las carcasas y altas
tasas de mortalidad (Frandoloso & Resende de Macebo, 2022).
Las tasas de
morbilidad y mortalidad asociadas con la enfermedad de Glässer pueden variar
del 5 al 10%, dependiendo de la gravedad de la enfermedad y virulencia de la
cepa de G. parasuis implicada (Aragon et al., 2019). La enfermedad
generalmente inicia de forma súbita presentando síntomas tales como anorexia, fiebre,
apatía, estornudos, tos, disnea, cianosis, conjuntivitis, claudicación o signos
nerviosos, los lechones afectados pueden morir durante la forma aguda o
evolucionar a la forma crónica en la que se observan artritis crónica y
adherencias en las serosas. Las lesiones comúnmente observadas corresponden a
pleuritis, pericarditis, peritonitis, oftalmitis, poliartritis y meningitis con
exudado fibrinoso o serofibrinoso (Frandoloso & Resende de Macebo, 2022).
Se han descrito
15 serotipos (Ser) con diferentes grados de virulencia, siendo considerados de
alta virulencia a Ser1, Ser5, Ser10, Ser12, Ser13 y Ser14; moderada virulencia
a Ser2, Ser4, Ser8 y Ser15, y baja virulencia a Ser3, Ser6, Ser7, Ser9 y Ser11.
Además, actualmente existe un porcentaje de aislados que no se pueden
serotipificar (Olvera et al., 2012; Pires Espíndola et al., 2019; Schuwerk et
al., 2020).
De esta manera
resulta clave la identificación de las serovariedades asociadas con brotes
regionales para el desarrollo de estrategias preventivas en cada región
productora de cerdos, que permitan el desarrollo de vacunas que contengan
antígenos para los serovares locales más prevalentes (Pires Espíndola et al.,
2019), lo que a su vez ayuda a limitar el uso de antimicrobianos en la
producción porcina. Estudios epidemiológicos en regiones productoras de cerdos
de varios países demuestran la circulación de un grupo heterólogo de serotipos,
siendo los serotipos 4 y 5 los más prevalentes en la mayoría de los países con
alta producción porcina (Pires Espíndola et al., 2019). En Paraguay,
no hay estudios sobre la prevalencia y distribución de los serotipos de G.
parasuis circulantes en el país, conocimiento necesario para poder plantear
estrategias de control y manejo de esta especie.
Materiales y métodos
Muestreo. El muestreo incluyó cinco granjas productoras de porcinos
ubicadas en el departamento de Itapúa desde el mes de febrero del 2022 a marzo
del 2023 (Figura 1). Se identificaron cerdos con síntomas respiratorios,
incluyéndose un total de 62 animales. En la figura 1, se observa la geoposición
de las granjas incluidas en el trabajo. En animales vivos se aplicaron métodos
de sujeción adecuados, las fosas nasales fueron higienizadas y las muestras
fueron colectadas con hisopos estériles en medio de transporte semisólido
Stuart. Para animales muertos se colectaron muestras de pulmón o de cornetes
durante las necropsias realizadas a campo, antes de las cinco horas post
mortem. La cantidad de muestras por granja se especifica en la Figura 2.
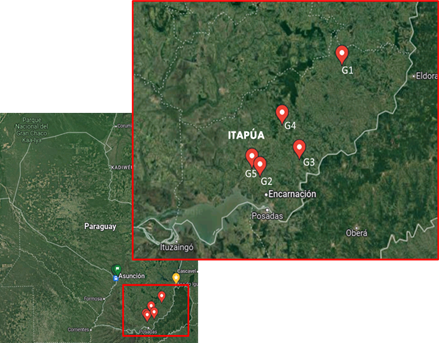
Figura 1. Ubicación geográfica de las granjas incluidas en el estudio.
Cultivo y
aislamiento de microorganismos. Las muestras fueron colectadas en las granjas y transportadas al
laboratorio, sembradas en Agar columbia Chocolate + NAD e incubadas por 48 hs a
37°C en atmósfera de microaerofilia. Se realizó tinción de Gram a las colonias
sospechosas, posteriormente se realizaron pruebas bioquímicas como Indol, Urea
y Test de catalasa (Frandoloso & Resende de Macebo, 2022).
Extracción de
ADN. Para la
extracción de ADN se tomaron colonias crecidas en placa y fueron suspendidas en
200 μL de solución PBS, se agitó con vórtex, y posteriormente se utilizó el kit
de purificación GeneJET™ Viral DNA/RNA (ThermoFisher Scientific) siguiendo las
instrucciones del fabricante (Thermoscientific, 2021).
Tipificación
molecular y virulencia. Para realizar la tipificación molecular se utilizaron los
cebadores descritos por Howell (2015), con modificaciones hechas por Lacouture
et al. (2017). Cada reacción de PCR consistió en 12,5 μL de 2x GoTaq® G2
Colorless Master Mix (Promega); 1 mM de cada uno de los cebadores, csp de agua
libre de nucleasas y 2 μL de ADN extraído, con un volumen final de 25 µL. Las
condiciones de ciclado fueron las mismas descritas por Lacouture et al.,
(2017). Para identificar la presencia de genes relacionados a virulencia se
utilizó la metodología descrita por Galofré-Mila et al., (2017) para la
amplificación de genes de virulencia vtaA.
Análisis de los
datos: Se clasificaron los
resultados teniendo en cuenta la granja de procedencia, los mismos fueron
analizados con estadística descriptiva, utilizando el Software RStudio
2022.07.0.
Resultados y discusión
Se identificó la presencia de G. parasuis en las 5 granjas incluidas
en este estudio (Figura 2). Un total de 12 cepas fueron aisladas,
identificándose el serotipo al que pertenecen el 50% (6/12) de los aislados,
perteneciendo a cuatro de los 15 (Ser2, Ser3, Ser7 y Ser8) serotipos
reconocidos, no correspondiendo los demás aislados a ningún serotipo conocido
actualmente, identificándose como NT (No Tipificables, Figura 2 y 3),
correspondiéndose con lo reportado para otros países (Pires Espíndola et al.,
2019; Schuwerk et al., 2020), lo cual estaría indicando la posible existencia
de más serotipos que los 15 únicos reportados actualmente, evidenciando la
necesidad de estudiar estas cepas NT. No se logró verificar la presencia de los
serotipos 4 y 5, considerados como los más prevalentes en la mayoría de los
países con alta producción porcina (Pires Espíndola et al., 2019).
Es importante destacar que el 80,65% de las muestras sintomáticas, fueron
ocasionadas por otras etiologías (OE), causadas por otros agentes. G. parasuis es una especie bacteriana
heterogénea que comprende cepas con amplias diferencias en virulencia. Esta
dualidad debe tenerse en cuenta a la hora de diagnosticar y controlar la
enfermedad (Costa-Hurtado et al., 2020). Es importante destacar que, prácticamente
cada granja presentó un serotipo específico, pudiendo esto relacionarse a la
procedencia de los cerdos, como lo reportado en otros países productores (Pires
Espíndola et al., 2019). Considerando la alta diversidad antigénica intrínseca
de las diferentes serovariedades de este microorganismo, es imprescindible
caracterizar minuciosamente las cepas envueltas en los casos para desarrollar
vacunas adecuadas que a su vez ayuden a reducir la utilización de
antimicrobianos de forma preventiva y disminuyan las probabilidades de crear
resistencias en cepas clínicas como ya ocurre en Brasil (Frandoloso y Resende
de Macebo, 2022).
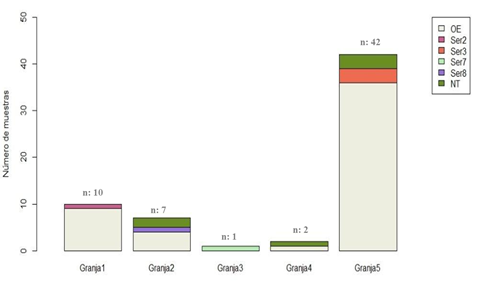
Figura 2.
Distribución de muestras y serotipos encontrados en cada una de las granjas. OE:
Otra etiología, G. parasuis: Ser2: Serotipo 2, Ser3:
Serotipo 3, Ser7: Serotipo 7, Ser8: Serotipo 8, NT: No
tipificable.
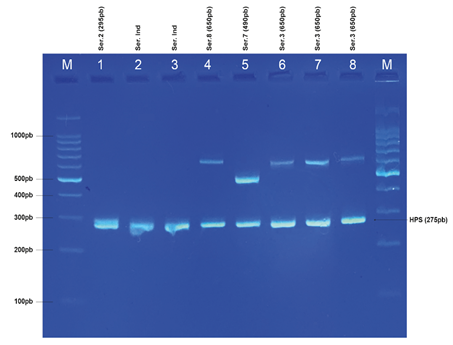
Figura 3. Perfil de
la PCR multiplex para cada uno de los aislados de G. parasuis
encontrados. Agarosa al 2%. M: marcador molecular; 1-8: muestras.
En cuanto a la identificación de genes
relacionados a virulencia, los resultados indicaron que la amplificación del
gen VtaA dio positivo en 11 de 12 de los aislados bacterianos. Aquellas que
fueron positivas se obtuvieron de muestras de pulmón (9/11, 82%), cornetes
(1/11, 9%) e hisopado nasal (1/11, 9%). La única cepa que dio negativo para el
gen de virulencia se obtuvo de una muestra de hisopado nasal. Estos resultados
sugieren que la mayoría de los aislamientos estudiados positivos al gen VtaA
pueden presentar mecanismos de resistencia al suero y a la fagocitosis. Estos
dos mecanismos se han reconocido como importantes factores de virulencia en la
enfermedad causada por G. parasuis (Cerdà-Cuéllar & Aragon, 2008;
Olvera et al., 2012).
De esta manera,
se logró identificar la presencia y serotipificar los aislados de G.
parasuis en cerdos con síntomas respiratorios procedentes de 5 granjas
dedicadas a la producción porcina en Itapúa, Paraguay. Si bien los serotipos
circulantes están clasificados dentro de los grupos de moderada y baja
virulencia todos fueron aislados de animales sintomáticos, y en casi el total
de los 12 aislados fue identificado el gen de virulencia VtaA.
De esta manera,
este trabajo se convierte en el primer reporte de serotipos de G. parasuis en
Itapúa-Paraguay, conocimiento fundamental para el control y manejo de este
patógeno.
Contribución de autores
Concepción
del estudio: N.D.Z.P., C.E.P.S., E.A.N.E. Diseño del
experimento: N.D.Z.P., L.N.T.S., C.E.P.S., E.A.N.E. Ejecución del
experimento: N.D.Z.P., Y.M.L., Y.D.S.L. Verificación del
experimento: N.D.Z.P., Y.D.S.L.Análisis/interpretación
de datos:
N.D.Z.P.,
Análisis
estadísticos: N.D.Z.P., L.N.T.S. Preparación del
manuscrito:
N.D.Z.P.,
Y.M.L., L.N.T.S. Edición
y revisión del manuscrito: L.N.T.S., C.E.P.S., E.A.N.E. Aprobación de la
versión final del manuscrito: L.N.T.S., C.E.P.S., E.A.N.E.
Fuente
de Financiamiento. Sin financiamiento externo.
Referencias Bibliográficas
Aragon, V., Segalés, J. &
Tucker, A. W. (2019). Glässer’s Disease. In Diseases
of swine (pp. 844–853).
Cerdà-Cuéllar, M. &
Aragon, V. (2008). Serum-resistance in Haemophilus
parasuis is associated with systemic disease in swine. Veterinary
Journal, 175(3), 384–389. https://doi.org/10.1016/j.tvjl.2007.01.016
Costa-Hurtado, M.,
Barba-Vidal, E., Maldonado, J., & Aragon, V. (2020). Update
on Glässer’s disease: How to control the disease under restrictive use of
antimicrobials. Veterinary Microbiology , 242
https://doi.org/10.1016/j.vetmic.2020.108595
Frandoloso, R., & Resende
de Macebo, N. (2022). Doença de Glässer. In R. M. C. Guedes & D. Barcellos
(Eds.), Doenças dos Suínos (Tercera, pp. 196–207).
Galofré-Milà,
N., Correa-Fiz, F., Lacouture, S., Gottschalk, M., Strutzberg-Minder, K.,
Bensaid, A., Pina-Pedrero, S., & Aragon, V. (2017). A robust PCR for the
differentiation of potential virulent strains of Haemophilus parasuis. BMC
Veterinary Research, 13(1). https://doi.org/10.1186/s12917-017-1041-4
Howell,
K. J., Peters, S. E., Wang, J., Hernandez-Garcia, J., Weinert, L. A., Luan, S.
L., Chaudhuri,
R. R., Angen,
Aragon, V., Williamson, S. M., Parkhill, J., Langford, P. R., Rycroft, A.N.,
Wren, B. W., Maskell, D. J., Tucker, A. W. & Consortium, D. J.
(2015). Development of a multiplex PCR assay for rapid molecular serotyping of Haemophilus
parasuis. Journal of clinical microbiology, 53(12), 3812-3821.
Lacouture,
S., Rodriguez, E., Strutzberg-Minder, K. & Gottschalk, M. (2017). Canada:
Serotyping of Haemophilus parasuis field isolates from disease pigs in
Quebec by indirect hemagglutination assay and multiplex polymerase chain
reaction (PCR). The Canadian Veterinary Journal, 58(8), 802.
Olvera,
A., Pina, S., Macedo, N., Oliveira, S., Aragon, V. & Bensaid, A. (2012).
Identification of potentially virulent strains of Haemophilus parasuis
using a multiplex PCR for virulence-associated autotransporters (vtaA). Veterinary
Journal, 191(2), 213–218. https://doi.org/10.1016/j.tvjl.2010.12.014
Pires
Espíndola, J., Balbinott, N., Trevisan Gressler, L., Machado, G., Silene Klein,
C., Rebelatto, R., Gutiérrez Martín, C. B., Kreutz, L. C., Schryvers, A. B.
& Frandoloso, R. (2019). Molecular serotyping of clinical strains of Haemophilus
(Glaesserella) parasuis brings new insights regarding Glässer’s disease
outbreaks in Brazil. PeerJ,
7, e6817. https://doi.org/10.7717/peerj.6817
Schuwerk, L., Hoeltig,
D., Waldmann, K. H., Strutzberg-Minder, K., Valentin-Weigand, P. & Rohde,
J. (2020). Serotyping and pathotyping of Glaesserella
parasuis isolated 2012–2019 in Germany comparing different PCR-based
methods. Veterinary Research, 51(1). https://doi.org/10.1186/s13567-020-00862-1
Thermoscientific.
(2021). GeneJET Viral DNA RNA Purification User Guide.
Fe de erratas
En el artículo “Glaesserella parasuis: serotipos y virulencia de
cepas aisladas de cerdos en Itapúa-Paraguay entre febrero del 2022 a marzo del
2023”, con número DOI: https://doi.org/10.57201/IEUNA2313373,
publicado en la Revista investigaciones y estudios-UNA , 14(1):84-89.
Los autores se disculpan sinceramente por el error involuntario en
la composición tipográfica en la página 87, sección Materiales y Métodos.
El contenido completo de dicha sección debe ser el siguiente, la
inclusión se visualiza en negrita:
Tipificación molecular y virulencia. Para realizar la
tipificación molecular se utilizaron los cebadores descritos por Howell (2015),
con modificaciones hechas por Lacouture et al. (2017). Fueron utilizadas
tres mezclas de cebadores que se detallan a continuación: PM1: funB (Ser1),
glyC (Ser3), wciP (Ser4), funQ (Ser7), funAB (Ser14); PM2: wzx (Ser2), funV
(Ser9), gltP (Ser13), funI (Ser15) y PM3: wcwK (Ser5/12), gltI (Ser6), scdA
(Ser8), funX (Ser10), amtA (Ser11). Cada reacción de PCR consistió en 12,5
μL de 2x GoTaq® G2 Colorless Master Mix (Promega); 1 mM de cada uno de los
cebadores, csp de agua libre de nucleasas y 2 μL de ADN extraído, con un
volumen final de 25 µL. Las condiciones de ciclado fueron las mismas descritas
por Lacouture et al., (2017). Para identificar la presencia de genes
relacionados a virulencia se utilizó la metodología descrita por Galofré-Mila
et al., (2017) para la amplificación de genes de virulencia vtaA.
https://doi.org/10.57201/ieuna2313937
![]() , , Universidad
Nacional de Asunción. Centro Multidisciplinario de Investigaciones Científicas
y Tecnológicas (CEMIT). San Lorenzo, Paraguay
, , Universidad
Nacional de Asunción. Centro Multidisciplinario de Investigaciones Científicas
y Tecnológicas (CEMIT). San Lorenzo, Paraguay![]() , Centro de
Desarrollo e Innovación Tecnológica (CEDIT). Hohenau, Paraguay
, Centro de
Desarrollo e Innovación Tecnológica (CEDIT). Hohenau, Paraguay![]()
![]() , Yasmine
Maluff Ladan1
, Yasmine
Maluff Ladan1![]() , Liliana
Noelia Talavera Stefani1,2
, Liliana
Noelia Talavera Stefani1,2![]() , Yanina Dionisia
Sapper Lacy1
, Yanina Dionisia
Sapper Lacy1![]() , Carolina
Elizabeth Prendeski Storaluk1
, Carolina
Elizabeth Prendeski Storaluk1![]() , Eliane
Aya Nishii Encina1*
, Eliane
Aya Nishii Encina1*![]()



